Каждые два года количество транзисторов, размещаемых на интегральной микросхеме, увеличивается вдвое. Так звучит каноническая версия закона Мура, – эмпирического наблюдения одного из основателей Intel, которое известно каждому, интересующемуся новостями технологий.
Как драйвер невероятного успеха IT-индустрии закон Мура давно стал, пожалуй, самым наглядным маркером прогресса. Однако есть области технологий, где даже эта «икона сингулярности» выглядит довольно бледно. Речь идет о технологиях чтения ДНК.
Первый человеческий геном, полученный в рамках десятилетнего международного проекта, обошелся примерно в три миллиарда долларов — это стоимость всей программы, включающая многочисленные научные исследования, который приходилось проводить по ходу работы. На момент окончания проекта (черновая версия генома была опубликована в 2001 году) стоимость прочтения еще одного генома сравнимого размера оценивалась приблизительно в 100 миллионов долларов. Нетрудно подсчитать, что сейчас, 14 лет спустя, если бы закон Мура действовал в биотехе, стоимость секвенирования составила бы около 750 тысяч долларов. На самом деле по состоянию на 2015 год стоимость чтения полного человеческого генома составляет около пяти тысяч долларов, а цена генотипирования — анализа только избранных, «ключевых» участков генома — опустилась уже до сотни долларов. Даже с учетом подготовки проб и логистики оба варианта процесса занимают уже не годы, а примерно несколько недель.
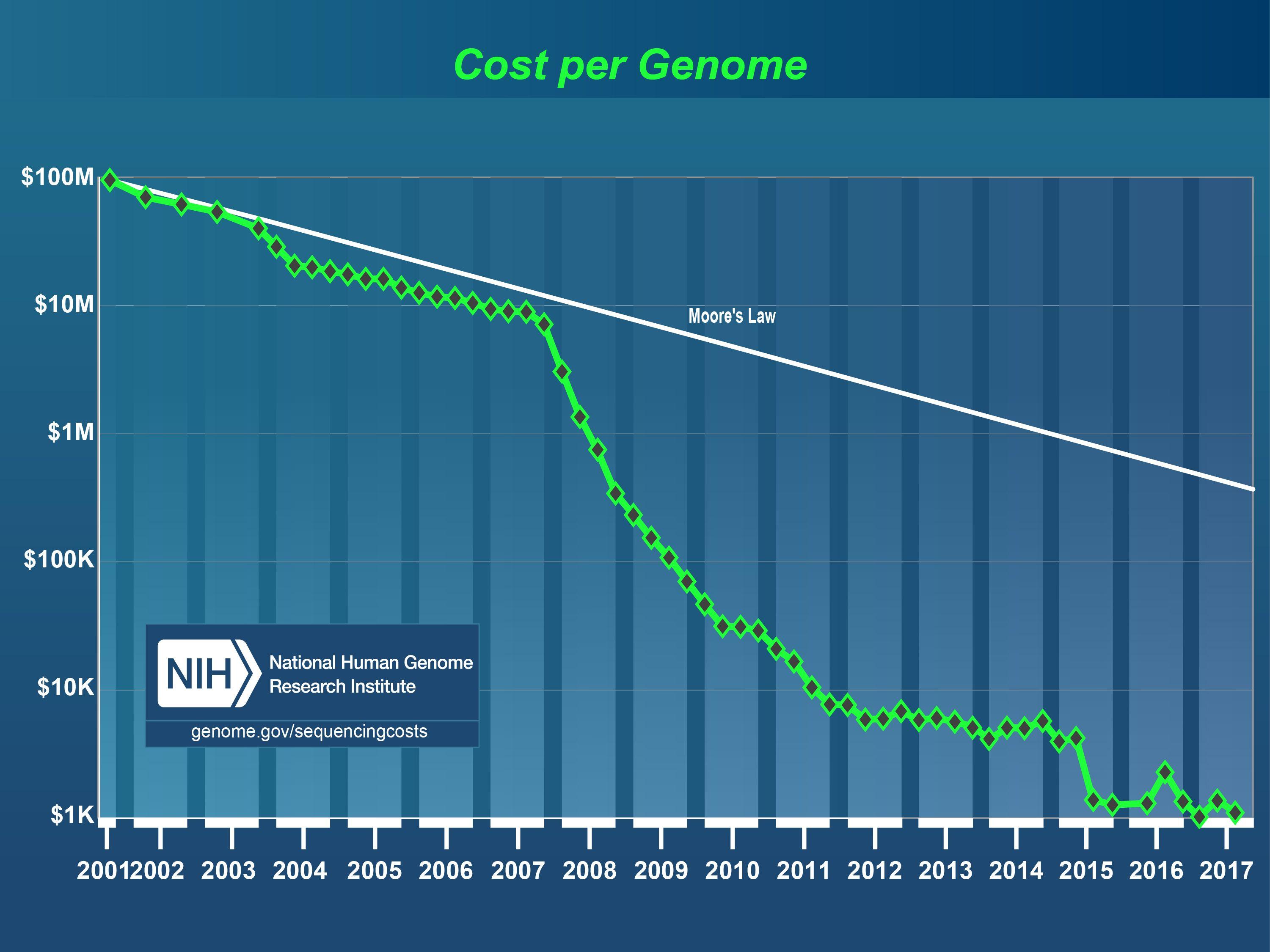
И тут возникают как минимум два вопроса. Во-первых, — что же произошло в технике секвенирования за это время, что позволило так радикально снизить стоимость секвенирования? И, во-вторых, почему мы на себе практически не замечаем этого взрывного прогресса?На второй вопрос ответить проще: «взрыв» геномной революции действительно произошел, но до нас, обычных потребителей, пока не дошла его «взрывная волна». Геномные данные уже давно стали радикально доступнее ученым и фармкомпаниям, но в жизнь самих их обладателей геномные данные стали выходить только в последние несколько лет. А вот первый вопрос — как и почему это произошло, как менялась технология секвенирования и как сегодня ученые читают ДНК — требует отдельного разбора.
Для начала нужно определится с тем, что такое секвенирование. Секвенированием (от слова sequence — последовательность) называют определение порядка элементраных единиц мономеров в полимере. Причем эти полимером может быть не только ДНК или РНК, но и, например, белок или даже полисахарид. Сам термин «секвенирование» возник в тот момент, когда стало понятно, что в биологии свойства и функции полимеров не могут определяться просто их составом, как привыкли думать химики: слишком похож оказался этот состав у совершенно разных с функциональной точки зрения молекул. А если дело не в составе, значит ключевую роль играет именно последовательность мономеров — идея, которая кажется тривиальной сейчас, но в 40-е годы прошлого века была совершенно новой.
Приоритет в открытии этой ключевой для биологии концепции принадлежит британскому ученому Фредерику Сенгеру. «Папа секвенирования» родился в 1918 году в семье врача и прожил очень длинную и исключительно плодотворную научную жизнь. Единственный в мире дважды нобелевский лауреат по химии (1958 и 1980 годов) он в полной мере успел застать геномную революцию, созданную прежде всего своими же руками.
Однако объектом первого в мире секвенирования, которое в конце 40-х провел Сенгер, была вовсе не ДНК или РНК. Это был инсулин – единственный в то время пептид, доступный в более-менее чистом виде в достаточных количествах. То, какие аминокислоты входят в состав инсулина было к тому моменту известно, но ученые полагали, что пропорции этих аминокислот в инсулине приблизительны, да и не слишком важны — предполагалось, что при создании белков жизнь руководствуется «аналоговым» подходом, «насыпая» аминокислоты в разные белки на глазок.
Именно Сенгер показал, что это не так. Ему удалось найти реагент, который избирательно взаимодействует только с одной аминокислотой из всей полипептидной цепочки: той, которая находится в самом начале пептида, и у которой, из-за этого, есть уникальная химическая группа. И если для исходного полипептида такая аминокислота одна, то после частичного разрезания концевой может стать любая из аминокислот, а значит все их можно идентифицировать. Путем разбивания инсулина на мелкие фрагменты и определения концевых аминокислот Сенгеру удалось (всего-то за восемь лет кропотливой работы) собрать полный «пазл» мономеров, и определить, таким образом, точную структуру инсулина. За эту работу Сенгер спустя всего шесть лет после последней публикации получил Нобелевскую премию по химии.
Однако после успеха с инсулином никто — ни Сенгер, ни его конкуренты, даже не пытались подступиться к секвенированию ДНК. «Самая главная молекула» выглядела слишком огромной и устрашающей для того, чтобы можно было попытаться прочитать ее последовательность. Кроме того, в каждой клетке, как известно, содержится всего одна или две копии геномной ДНК, а значит получить достаточное количество одинаковых молекул (чего требует метод), довольно сложно.
Тактической целью стала другая нуклеиновая кислота, РНК. Точнее говоря, ее отдельная разновидность, транспортная РНК, которая в клетке используется в качестве адаптера, «подносящего» аминокислоты при синтезе белка. Ее длина составляет всего 70-80 нуклеотидов, а число копий на клетку достигает сотен тысяч штук. Для секвенирования РНК Сенгер применил ту же общую стратегию: мечение концевого мономера с последующим частичным разрушением молекулы на множество фрагментов. Однако тут удача ему изменила. Роберт Холли с соавторами из Корнельского университета смог опубликовать последовательность одной из тРНК дрожжей уже в 1965 году, именно Холли стал первым человеком, определившим последовательность какой-либо нуклеиновой кислоты. И хотя всего три года спустя группе Сенгера удалось секвенировать РНК почти вдвое длиннее (это была одна из тех молекул, что входят в состав рибосомы), по-настоящему отыграться за это поражение ему удалось только спустя десять лет.Метод секвенирования ДНК, несущий сейчас имя Сенгера, был опубликован в 1977 году. На протяжении более 30 лет, вплоть до середины нулевых годов, он оставался главным способом определения последовательности любой нуклеиновой кислоты: именно этим методом (с незначительными модификациями) был прочитан геном человека. И до сих пор секвенирование по Сенгеру является самым точным методом, к которому обращаются при необходимости проверить результаты секвенирования нового поколения.
Идея, лежащая в основе секвенирования по Сенгеру, настолько проста и изящна, что на ней хочется остановится подробнее. До сих пор, как мы видели, все методы чтения последовательности были основаны, условно говоря, на разрушении: на получении чистого вещества, его фрагментировании и восстановлении «пазла» из получившихся фрагментов. Сенгер решил действовать противоположным образом: читать ДНК не фрагментированием, а синтезом, причем использовать для этого природный фермент, ДНК полимеразу — ту самую молекулярную машину, которая удваивает ДНК перед делением клетки.
Процедура проводится следующим образом: фрагмент ДНК, помеченный с одного из концов радиоактивным изотопом, разделяют на четыре пробирки. В каждую из них добавляют реактивы, необходимые для синтеза новой ДНК, в том числе одиночные «буквы», которые полимераза должна будет связать в новую нить — в точном соответствии с исходной матрицей. Однако помимо обычных «букв» к раствору добавляется некоторое небольшое число специально «испорченных» — таких, после которых невозможно присоединить следующую «букву» (они просто лишены соответствующего места для связи).
В результате в конце синтеза в каждой пробирке появляется набор ДНК разной длины, причем каждая из молекул несет радиоактивную метку в начале и испорченную «букву» на конце. Поскольку в каждую из четырех пробирок мы кладем только один вид испорченных оснований, мы знаем, какой буквой кончаются все фрагменты ДНК в данной пробирке. Теперь достаточно разогнать содержимое всех четырех пробирок в геле, разделяющем ДНК по длине и приложить к нему фотопленку, которая зафиксирует скопления радиоактивности. На пленке появится «лестница» из ступенек, каждая из которых будет соответствовать одной букве в последовательности. Поднимаясь по лестнице, мы прочтем всю последовательность исходной ДНК.
Метод Сенгера оказался исключительно надежным и удобным для чтения последовательностей. Возможно, именно благодаря тому, что он полагался на «выдрессированные» в ходе миллиардов лет эволюции природные ферменты, а не синтетические реактивы. Именно с помощью этого метода (точнее, с помощью его непосредственного варианта-предшественника) Сенгеру удалось прочитать первый в истории полный геном отдельного организма — геном бактериофага ϕX174, содержащий всего 5386 оснований (кстати, этот же фаг в 2003 году стал первым организмом, геном которого был полностью синтезирован искусственно).Уже несколько лет спустя, в середине 80-х, когда инкрементные улучшения постепенно увеличивали скорость и мощность секвенирования, стали появляться полные геномы все более сложных вирусов и ученые впервые заговорили о возможном секвенировании геномов высших организмов, в том числе и человека.
Подобраться к этой задаче стало возможно тогда, когда появились методы молекулярного клонирования, позволявшие вырезать из генома отдельные фрагменты, затем вставлять их в модельные организмы (кишечную палочку или дрожжи), а потом постепенно, по кусочку, секвенировать – тем же самым методом Сенгера.
В 1990 году стартовала международная программа «Геном человека», в которой каждому коллективу из Америки, Европы и Японии были выделены отдельные участки предварительно размеченного генома для секвенирования. К этому моменту несколько биотехнологических компаний, прежде всего Applied Biosciences, научились автоматизировать процессы секвенирования по Сенгеру. Сначала секвенаторы могли самостоятельно только читать «лестницы» на фотопленке, превращая их в буквенную последовательность, а затем сам процесс разделения фрагментов ДНК удалось перенести из геля (который нужно было каждый раз заливать заново) в тонкий капилляр. Радиоактивные метки заменили флюоресцентными, и это позволило читать последовательность прямо во время прохода сквозь капилляр: четыре цвета — четыре разные буквы.
Одним из первых, кто осознал возможности автоматизации секвенирования, был Крейг Вентер — будущий одиозный зачинатель «гонки геномов», решивший опередить в прочтении генома человека всю международную коллаборацию (а также автор первого живого организма с полностью синтетической ДНК). В 1992 году, спустя два года после старта проекта «Геном человека», Вентер организовал собственный институт с броской аббревиатурой TIGR (The Institute for Genomic Research), в котором секвенирование ДНК было впервые поставлено на поток. В 1995 году группе Вентера удалось получить первый геном полноценного клеточного организма, точнее даже двух: бактерий Haemophilus influenzae и Mycoplasma genitalium.
С чисто биологической точки зрения принципиальным было то, что речь шла именно о полноценных клеточных геномах: вирусы живут только в клетках и в своей жизни почти во всем полагаются на клеточные системы, кодируемые не собственным геномом, а геномом клетки. И только последние содержат полную необходимую для жизни информацию и поэтому гораздо интереснее вирусных геномов.С технической точки зрения новаторство Вентера заключалось в том, что он впервые применил радикальный подход к секвенированию генома. Как уже говорилось, до сих пор ученые работали с геномом «по кусочкам», что позволяло читать ДНК самых сложных организмов но и требовало огромных затрат времени на клонирование. Вентер стал пионером так называемого «метода дробовика», когда весь геном целиком нарезается на множество коротких пересекающихся фрагментов, которые прочитываются, а затем собираются снова: снизу вверх.
Такой подход существенно упрощает все стадии, требующие участия человека и хорошо подходит для автоматизации. Однако у него есть два принципиальных недостатка. Во-первых, сборка генома из миллионов и миллионов коротких фрагментов требует огромных вычислительных ресурсов, подразумевает создание принципиально новых математических алгоритмов и требует многократного покрытия (например, чтобы собрать геном длиной 100 нуклеотидов вам нужно прочитать фрагментов общей длиной в 1000 — в этом случае говорят про десятикратное покрытие).
А во-вторых, работает этот метод по принципу «пока не сделано всё — не сделано ничего». Именно благодаря этой особенности наблюдать за гонкой геномов было особенно интересно: если бы качества и количество «сырых» последовательностей Вентеру немного не хватило, вся авантюра c секвенированием оказалась бы пустой тратой времени. Этого, однако, не случилось. Оба генома — и полученный в ходе международного проекта и собранный частной компанией Вентера были опубликованы в двух выпусках Nature и Science, вышедших на одной неделе в феврале 2001 года.
Услышав про проект «Геном человека» многие спрашивают: — а какого именно? Кем был тот человек, на прочтение генома которого было потрачено столько денег и усилий ученых? И хотя ответ тривиален (никем он не был, это референтный геном, ДНК для которого была получена у нескольких анонимных доноров) такой вопрос бьет в самую точку. Никто из нас не является обладателем консенсусного генома, в ДНК каждого существует огромное количество уникальных отличий, и именно эти отличия делают нас теми, кто мы есть. Поэтому в первый же день после публикации референтного генома человека стало ясно, что главной целью всех последующих исследований в человеческой геномике станут индивидуальные отличия.
Есть определенная ирония в том, что первыми людьми, персональный геном которых был прочитан, стали два главных соперника «гонке геномов»: первооткрыватель структуры ДНК Джеймс Уотсон и уже знакомый нам Крейг Вентер. По сравнению с референтным геномом у каждого из них было обнаружено около трех миллионов индивидуальных полиморфизмов — однобуквенных замен, которые отличаются от человека к человеку. Стоимость секвенирования обоих индивидуальных геномов к моменту их прочтения в 2007 году составила около миллиона долларов. И это, конечно существенно ниже, чем 100 миллионов в 2001 году, но все равно немало: с такими ценами рассчитывать на прочтение геномов сотен или тысяч человек или предлагать такую услугу обычным людям было бы странно. Однако, к счастью, как раз в тот момент, когда референтный геном был создан, созрела технология, позволяющая «вылавливать» индивидуальные отличия в геномах принципиально проще и дешевле обычного секвенирования. Речь идет о технологии ДНК-микрочипов.
ДНК-микрочип — это небольшая пластинка, на которой с помощью технологии, напоминающей фотолитографию, закреплены короткие одноцепочечные фрагменты ДНК. При инкубации с раствором образца две молекулы ДНК — одна на чипе, другая в образце — могут образовать прочную пару. Если все молекулы образца предварительно пометить флюоресцентным маркером, то после инкубации мы увидим на чипе светящиеся точки в тех местах, где образовались прочные пары. И если это произошло, значит последовательности фрагментов ДНК в образце точно совпали с последовательностями на чипе — а их мы знали заранее.Установив, какие в популяции существуют индивидуальные особенности, мы можем создать микрочип с сотнями тысяч различных полиморфизмов. Это позволит получать информацию о наличии тех или иных «однобуквенных» замен в вашей ДНК всего за несколько часов. Формально, такая процедура не является секвенированием, но она позволяет читать последовательности ДНК, варианты который мы уже знаем. Использовать микрочипы для чтения совершенно новых последовательностей нельзя (хотя работы в этом направлении ведутся), но когда речь идет о персональной геномике, этого и не требуется: ДНК разных людей, как известно, совпадают на 99 процентов. С помощью современных микрочипов можно прочитать около миллиона известных полиморфизмов, то есть примерно одну треть от того их количества, которое присутствует в геноме.
ДНК-микрочипы стали появляться в научных лабораториях в 90-х, а в середине 2000-х появились первые компании, предлагающие анализ персонального генома на их основе. Небезызвестная 23andMe, основанная бывшей женой Сергея Брина, как раз была одной из первых таких компаний. Сейчас у компании Энн Вожитски появилось множество конкурентов, причем, как в мире, так и в России.
Однако сегодня технологиям генотипирования наступают на пятки (и по скорости, и по стоимости) так называемые методы секвенирования нового поколения. Именно их появление обвалило стоимость процедуры с миллионов до тысяч долларов. Это методы очень разные, и обо всех них рассказать не получится.
Немного остановится можно, пожалуй, только на так называемом пиросеквенировании — методе, который основан на гидролизе пирофосфата. При соединении нуклеотидов друг с другом в цепочку ДНК в раствор всегда выбрасывается это соединение — высокоэнергетичный фрагмент нуклеотида, который затем бесследно разрушается и своей «гибелью» обеспечивает однонаправленность реакции синтеза. В середине 2000-х многие научные группы независимо заметили, что разрушение пирофосфата уместно использовать при секвенировании: его можно «скармливать» специальному ферменту, который умеет превращать энергию связи пирофосфата в импульс света. Тогда, по наличию или отсутствию вспышки можно будет судить — прошла ли реакция присоединения нуклеотида к цепочке или нет. Когда на матрице ДНК образца синтезируется ее копия, вспышка означает наличие в нуклеиновой кислоте комплементарного основания.
Выглядит это так: ДНК режут на миллионы коротких фрагментов, наносят на микроскопические шарики, копируют их (так, чтобы на одном шарике были только идентичные копии одного фрагмента) и распределяют по микроскопическим ячейкам, сделанным в специальной подложке. После этого в ячейках начинается синхронная реакция. Сквозь подложку пропускают один вид нуклеотидов — если в ячейке при этом происходит вспышка, значит этот нуклеотид подходит для синтеза, значит на матрице находится комплементарный нуклеотид. Затем подложку отмывают от первого нуклеотида и подают второй — на этот раз загораются другие ячейки, те, в которых есть соответствующее комплементарное основание. Так, многократно промывая ячейки четырьмя нуклеотидами, биоинженеры читают последовательность ДНК по вспышкам в отдельных ячейках. Главная особенность этого и подобных методов — возможность проводить огромное количество параллельных реакций. И хотя точность реакции в каждой из ячеек невелика, огромное количество таких ячеек делает секвенирование очень быстрым и, следовательно, дешевым.И все же пока полногеномное секвенирование не может сравняться по стоимости с генотипированием. Да, в исследовательских лабораториях технологии нового поколения уже вытесняют генотипирование из традиционных для этого метода задач (например, для анализа РНК и экспрессии генов). Но вот в персональной геномике дела обстоят иначе: особенности генома, которые заметны только при полном секвенировании и не видны микрочипам настолько редки, что настоящее секвенирование кажется стрельбой из пушки по воробьям.
В последние год-два на рынке полногеномного чтения ДНК наблюдается небольшой застой (кстати, напоминающий ситуацию перед тем, как появились методы нового поколения). Поэтому можно ожидать, что в ближайшие годы потребительская геномика будет по-прежнему полагаться на ДНК-микрочипы. Учитывая, то, насколько доступными они уже стали, даже введение новых революционных методов секвенирования вряд ли что-либо сильно поменяет на потребительском рынке. А значит, наступил момент, когда дело уже не в технологиях, с помощью которых получаются геномные данные, а в их интерпретации. Но это уже совсем другой разговор.
Автор: Александр Ершов
