Быстрая, недорогая и высокоточная детекция нуклеиновых кислот теперь станет возможной с помощью метода SHERLOCK, основанного на работе системы CRISPR-Cas13a. Им можно будет пользоваться для выявления патогенов, генотипирования и мониторинга генетических заболеваний.
Ключевым элементом новой технологии является нуклеаза Cas13a, ранее известная как C2c2 — это фермент, который способен узнавать специфическую последовательность в нити РНК и после этого разрезать любые РНК, которые попались ему рядом. Об открытии Cas13a/C2c2 мы уже писали ранее. По своей функции эта нуклеза аналогична более известной Cas9, которая широко применяется для редактирования генома. Однако, в отличие от последней, Cas13a/C2c2 имеет два важных отличия: во-первых, она работает на уровне РНК, а не ДНК, а во-вторых, вносит в мишень не «хирургический точный» разрыв, а полностью ее уничтожает. Такие особенности делают ее неподходящей для редактирования генома, однако, как оказалось, их же можно успешно использовать в совсем других целях.
Разработанная на основе Cas13a/C2c2 система детекции нуклеиновых кислот позволяет работать с аттомолярными (10-18 моль на литр) концентрациями ДНК или РНК, и при этом быстро и точно выявлять разные штаммы вирусов, различать патогенные бактерии, генотипировать ДНК человека и определять опухолевые мутации во внеклеточной ДНК, циркулирующей в плазме крови.
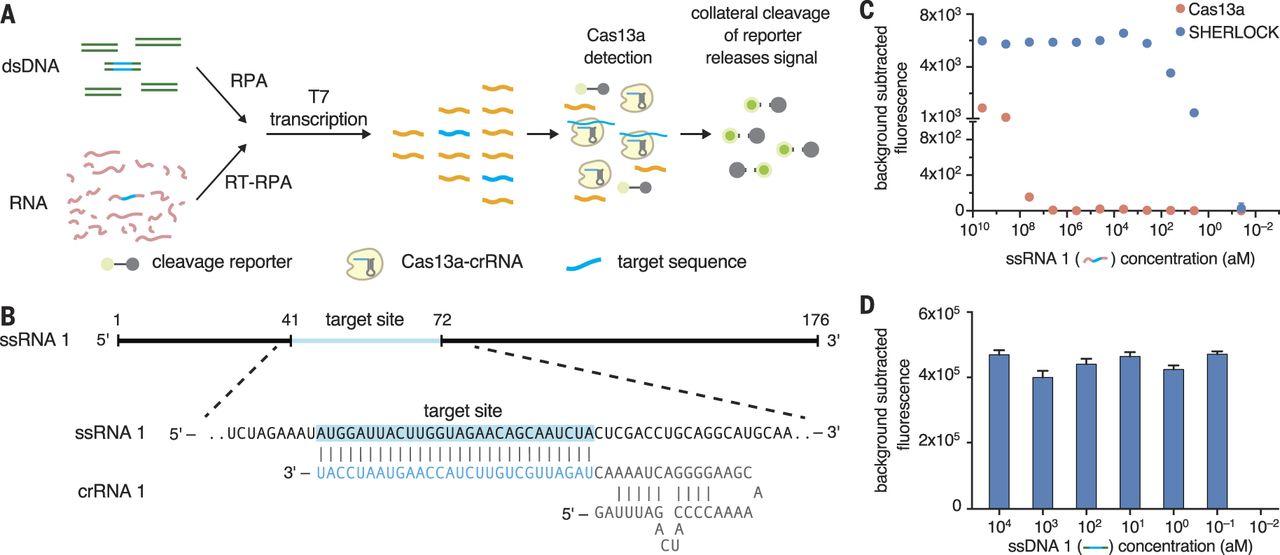
Для того, чтобы исследовать возможности этой системы, белок Cas13a бактерии Leptotrichia shahii инкубировали с целевой РНК (ssRNA 1), CRISPR РНК (crRNA) и репортерной РНК, которая позволяла убедиться, что фермент сработал. При этом в кассете CRISPR были закодированы фрагменты ssRNA 1, поскольку именно ей фермент пользуется как шпаргалкой для поиска целевой РНК. Система работала in vitro и показывала неплохую чувствительность, но чтобы добиться аттомолярной чувствительности эксперимента, ученым пришлось некоторое время подбирать условия предварительной амплификации (метода увеличения количества нуклеиновых кислот). Оптимальной работы фермента удалось достичь с помощью рекомбиназной полимеразной амплификации, увеличивающей число копий ДНК, после чего ДНК конвертировали в РНК, после чего ssRNA 1 детектировали с помощью Cas13a. Фермент чувствовал определенную последовательность ssRNA 1, сравнив ее с той, что закодирована в CRISPR, и резал окружающие РНК, в том числе репортерную. Разрезанная репортерная РНК начинала флуоресцировать, благодаря чему можно было убедиться, что фермент нашел цель. Этот комплекс методов назвали SHERLOCK (Specific High Sensitivity Enzymatic Reporter UnLOCKing).
Методы капельно-цифровой ПЦР подтвердили, что для работы этой системы достаточно всего одной молекулы исходной РНК или ДНК. Чувствительность SHERLOCK совпадает с чувствительности метода количественной ПЦР и капельно-цифровой ПЦР — двумя основными способами детекции нуклеиновых кислот, при этом его результаты при многократных повторах эксперимента показывают меньшую вариабильность.
Эффективность метода была протестирована на лентивирусах, содержащих геномные фрагменты вируса Зика и вируса лихорадки Денге. SHERLOCK распознавал вирусные частицы, концентрация которых не превышала двух аттомоль, и различал два вышеуказанных типа между собой. Выяснилось, также, что SHERLOCK можно подвергать высушиванию и последующему восстановлению, и его чувствительность при этом не падает ниже 20 аттомоль. Детекцию при этом можно проводить на стекловолокнистой бумаге, что делает метод удобным для использования практически в любых условиях.
Действие SHERLOCK проверяли на образцах мочи, слюны и сыворотки крови, где он успешно выявлял нуклеиновые кислоты с концентрацией около трех аттомоль. При этом нуклеиновые кислоты не обязательно было подвергать предварительной очистке. SHERLOCK также правильно различает разные штаммы патогенных бактерий. Используя методы мутагенеза для внесения искусственных мутаций, ученые выяснили, что система позволяет детектировать даже однонуклеотидные замены (SNP). Это, в свою очередь, дает возможность проводить генотипирование человеческих геномов, которое основывается на их однонуклеотидных различиях. SHERLOCK успешно «опознал» четыре человеческих образца по пяти геномным локусам, взятых из базы данных 23andMe, причем мог детектировать как гомозиготные, так и гетерозиготные мутации. Последним этапом был поиск внеклеточных фрагментов ДНК с раковыми мутациями, свободно плавающими в образцах крови больного человека, детекция которых обычно затруднена из-за большого количества обычной ДНК. Выяснилось, что метод способен зафиксировать даже 0,1 процентную концентрацию нуклеиновой кислоты, несущей раковые мутации.
Ученые полагают, что применение SHERLOCK может стать важным этапом методов количественной оценки нуклеиновых кислот, мультиплексных экспрессионных анализов и других чувствительных методик, в том числе определения уровня контаминации образцов. Также его можно применять для детекции патогенных мутаций в живых клетках и аллелеспецифичной экспрессии. Один тест на стекловолокнистой бумаге при этом будет стоить всего порядка 61 цента.
Статья опубликована в Science.
Подробности о перспективах исследований CRISPR и истории открытия Cas13a/C2c2 можно узнать в недавнем интервью с одним из авторов этого открытия, Константином Севериновым.
Авторы: Анна Казнадзей, Александр Ершов
